KEGG数据库中文教程
博奥生物有限公司生物芯片北京国家工程研究中心北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:http://www.c

博奥生物有限公司
生物芯片北京国家工程研究中心
北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:http://www.capitalbio.com
KEGG 数据库中文教程
,
博奥生物有限公司
生物芯片北京国家工程研究中心
北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:http://www.capitalbio.com
快速导航
1. 这篇教程介绍了什么?
2. KEGG数据库里面有什么?
3. 我如何查询某一特定的代谢途径(pathway)的信息,例如Glycolysis / Gluconeogenesis?
4. 我如何查询某一化合物的信息, 例如Pyruvate?
5. 我如何查询Pyruvate 涉及了哪些生化反应?
6. 我如何查询某一基因的信息, 例如gltA ?
7. 我如何知道Bacillus subtilis是否有gltA?
8. 我如何查询 gltA 在其他物种中的同源基因?
9. 我如何列出某一代谢途径中涉及的所有的酶?例如cytrate cycle pathway(TCA 循环)
10. 我如何知道人类的cytrate cycle中pyruvate carboxylase这种酶有多少化合物与其发生相互作用?
11. 我如何查询人类由Citrate 生成Acetyl-CoA 的可能步骤?
12. 我有一条未知的序列,如何查询KEGG 数据库中是否有基因或酶与其对应?
,
博奥生物有限公司
生物芯片北京国家工程研究中心
北京市昌平区生命科学园路18号
邮编:102206
电话:86-10-80726868 传真:86-10-80726898 网址:http://www.capitalbio.com
一. 简介
代谢(Metabolism)是指细胞内发生的各种化学反应的总称。一个代谢途径(Metabolic
pathway )包括一系列互相联系的反应(reaction),反应中的酶( enzyme)以及反应中的前体或者产物(substrate) 。 随着现代生物信息学的进展,我们可以通过计算机来展示,以至预测细胞内的代谢途径。
现在常用的查询代谢途径的数据库主要包括:KEGG(http://www.genome.jp/kegg/), GenMAPP(), BioRag() 等。本教程主要介绍KEGG 数据库的使用方法。
KEGG (Kyoto Encyclopedia of Genes and Genomes)是由日本京都大学和东京大学联合开发的数据库,可以用来查询代谢途径,酶(或编码酶的基因),产物等,也可以通过BLA ST 比对查询未知序列的代谢途径信息。KEGG 主要通过Web 界面进行访问,同样也可以通过本地运行的Perl 或java 程序进行访问,使用很方便。
本教程将结合实例来介绍KEGG 数据库的使用方法,希望能您能通过本教程了解KEGG 数据库的基本使用方法。
二. 使用方法
1. 首页 ● ● ● ●
PATHWAY (代谢途径数据库),可以查询各种代谢途径。
BRITE (代谢通路及同源基因数据库),这个数据与PATHWAY 数据库不同的是,可以查询酶和底物之间的关系,也可以查询某种酶的同源基因。 GENES (基因数据库), 可以查询不同的基因或基因组的信息。 LIGAND (配体数据库), 可以查询反应中各种化合物的信息。
打开的首页,我们可以看到KEGG 提供的四个主要的数据库(图1箭头所示)。
,
博奥生物有限公司
生物芯片北京国家工程研究中心 北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:

图1. KEGG数据库首页
如果点击 KEGG2——KEGG Table of Contents 这个链接,则会出现一个类似于网站地图的页面,在这个页面上,我们可以查询各数据库的更新信息,也可快速访问KEGG 提供的各个数据库。
点击会列出KEGG 对各物种的代码。KEGG 使用三个英文小写字母来代表各个物种,例如人类Homo sapiens,代码是hsa 。再如小鼠Mus Musculus则是mmu 。
2. PATHWAY 数据库的使用
在首页上点击PA THWAY 的链接(或者先选择KEGG2,再在出现的表格中Database 选项下选择KEGG PATHWAY) 。您就会看到KEGG 收录的所有代谢途径信息(图2)。
,
博奥生物有限公司
生物芯片北京国家工程研究中心 北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:

图2. PA THWAY 数据库界面
例如如果想要查询Glycolysis / Gluconeogenesis这个代谢途径(图2)。
1) 从首页,或者KEGG2的表格界面中进入数据库
2) 点击Carbohydrate Metabolism中的
新页面即显示出此途径的信息(图3A )。方框中表示的是反应中的酶,例如2.7.1.41,这是酶的EC number ,国际酶学委员会的编号。小圆圈代表的是反应中的化合物,例如α-D-Glucose-1P 。箭头代表的是反应的方向。虚线表示此反应可以通过中间产物与其他途径发生联系。
或者您也可以通过Search PATHWAY for这个选项直接搜索Glycolysis / Gluconeogenesis的信息。搜索的结果中会显示出满足条件的所有物种的pathway 。
,
博奥生物有限公司
生物芯片北京国家工程研究中心 北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:
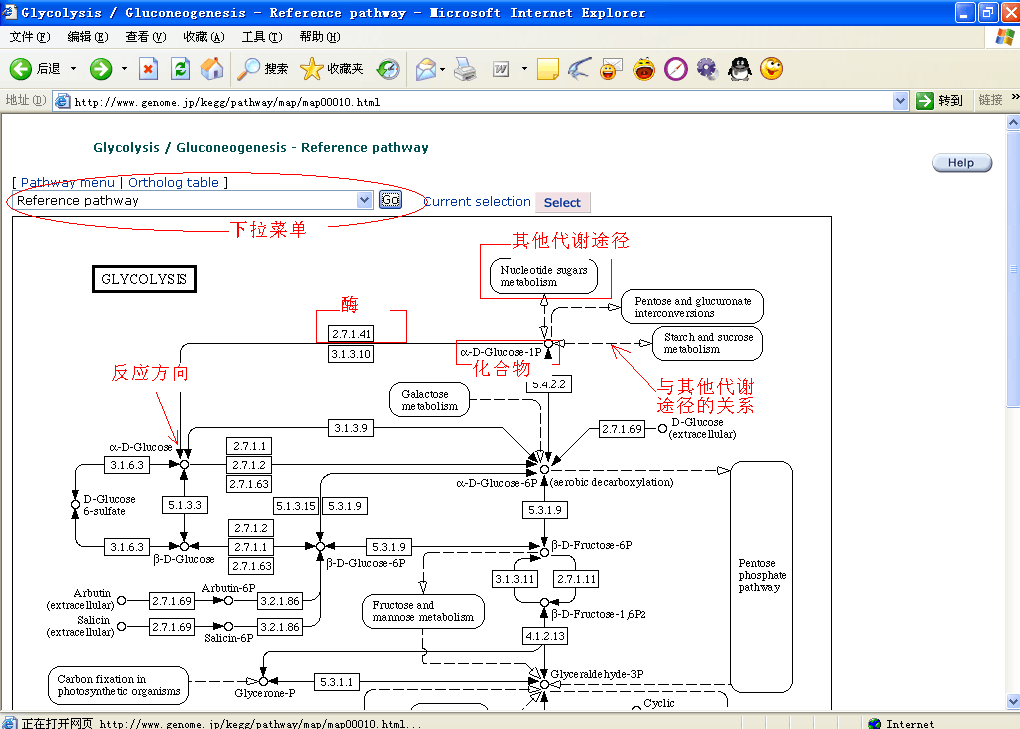
(A )

(B )
图3. 代谢途径图片信息
A :Glycolysis / Gluconeogenesis途径的全面信息 B:人类中Glycolysis / Gluconeogenesis途径的信息
如果想要知道人类的Glycolysis / Gluconeogenesis途径的具体信息。
,
博奥生物有限公司
生物芯片北京国家工程研究中心
2) 点击Go 北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:http://www.capitalbio.com 1) 点击此界面左上角的下拉菜单。选择人类 Homo Sapiens(human)
即可见图3B 的画面。绿色的方框代表人类含有这种酶,例3.1.3.9。你也可以通过下拉菜单旁的Select 按钮自行设置下拉菜单中显示的物种名称。
点击标有3.1.3.9的绿色方框,即可显示人类中此酶的信息(图4)。Entry 是KEGG 中此酶的ID 。Gene name是酶的简化名。Definition 包括酶的通用名称和EC number。KO 是在KEGG 数据中这种酶的同源序列号。Pathway 中列出了这种酶涉及的代谢途径。此外,还有一些其他的信息,例如编码这种酶的基因在基因组中的位置(Position ),编码酶的基因序列(NT seq)和蛋白序列(AA seq)等。

图4. 酶3.1.3.9的信息
3. LIGAND数据库的使用 LIGAND 数据库中,可以查询到化合物,反应或者参与反应的酶。可以在上面的搜索框中进行名称查询,也可以在下面的Ligand Relational Database里进行配体或反应关系的查询。
例如想要查询数据库中Pyruvate (丙酮酸盐)的信息(图5A )。
1) 在Search COMPAND的下拉菜单中选择Name
2) 输入Pyruvae
3) 点击Go
就会出现图5B 的画面。在给出的结果中可见它的entry number (C00022),分子结构,化学式等属性,也可以点击Entry number后进入另一个界面查看它更多的信息。
,
博奥生物有限公司
生物芯片北京国家工程研究中心 北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:


(A)
(B)
图5. LIGAND 数据库界面及Search Compound的使用
再例如,我们需要查询Pyruvate (丙酮酸盐)所涉及的化学反应的信息(图6A )。
1) 在Search REACTION的下拉菜单选择Reactant Entry
,
博奥生物有限公司
生物芯片北京国家工程研究中心
2) 输入Pyruvate 的entry number,C00022 北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:http://www.capitalbio.com
3) 点击Go 就会出现Pyruvate 所涉及的反应(图6B )。

(A )

(B )
图6. Search REACTION的使用
,
博奥生物有限公司
生物芯片北京国家工程研究中心
4.GENES 数据库的使用
北京市昌平区生命科学园路18号 邮编:102206 电话:86-10-80726868 传真:86-10-80726898 网址:http://www.capitalbio.com GENES 数据库可以用来查询基因及基因组的信息。
例如想要查询gltA (柠檬酸盐合成酶)这种基因的具体信息(图7A )。
1) 在Search 的下拉菜单选择Genes (默认,可不选)
2) 输入gltA
3) 点击Go
在出现的结果中即可查得所有编码这种酶的基因(图7B )。像是第一个结果eco:b0720,eco 是物种名(Escherichia coli K-12 MG1655), b0720是entry name。gltA 是基因名,之后还有基因的全称,以及编码的酶的EC number 等。物种名可以通过首页上的KEGG Organisms 来查询。
再例如我们想要查询Bacillus subtilis中是否有gltA 这种基因(图7C )。
1) 在首页的KEGG Organisms中找到Bacillus subtilis的缩写为bsu
2) Search Organism中输入bsu
3) 再输入gltA
4) 点击Go 即可查得相应信息

(A)
